转自
lncRNA功能和机制的研究,成为了现如今科学界的“新宠”。超高的关注率,说明在这个领域能发表一篇高水准的科学论文,需要付出更多的努力和超前的思路。今天,就为大家带来Nature Genetics的一篇关于lncRNA的文章,让小编给大家一步步解释作者是如何通过整合公共数据库的结果,验证了作者的科学假设,发表了大NG。
简单介绍
论文题目:Modulation of long noncoding RNAs by risk SNPs underlying genetic predispositions to prostate cancer
发表杂志:Nature Genetics
发表年份:2016年10月
影响因子:27.959
文章解读
这篇文章中,作者并没有自己进行测序,而是用一些新的思路和巧妙的构思,结合了前列腺癌(Prostate Cancer; PC)的SNP数据、表达数据、CHIP-Seq数据等,验证了与前列腺癌显著相关的lncRNA PCAT1,并且探索了PCAT1的调节机制,并且在生物学实验中做了验证。小编给大家分享这篇文章,是想大家可以借助作者的思路,在自己感兴趣的疾病中也能发现到一些感兴趣的现象。因为只凭借老的分析流程,把自己的文章推到一个好的杂志,还是有相当的难度的。闲言少叙,下面小编为大家梳理一下作者的工作流程。
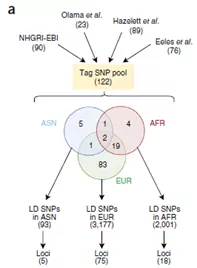
1. 作者从DNA入手,结合之前所发的文章以及GWAS catalog数据库 (http://www.ebi.ac.uk/gwas/)的结果,一共筛选出了122个和PC显著相关的SNP位点。由于担心无法找到真正的功能性位点,作者根据这122个SNP的连锁不平衡(LD > 0.8),一共找到了4989个SNP,进行之后的研究。
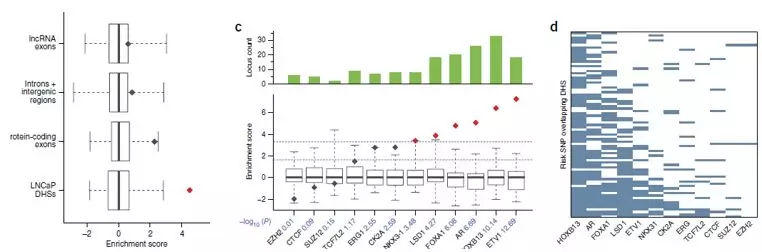
2. 找到了这些SNP,作者做了三次不同程度的富集分析,逐步的缩小了范围,增加了下一步研究的证据。首先,分析后发现这些SNP显著富集在DNA酶I超敏感位点(DNase I–hypersensitive sites;DHSs);之后,作者发现这些loci富集于和雄性激素受体(androgen receptor; AR)相关的一些转录因子,而且这些DHXs至少包含一个risk SNP,一半以上都和这些转录因子结合;最后,作者又发现这些位点富集于前列腺癌细胞系的H3K27ac、H3K4me1区域。这些证据证明了这些risk SNP主要是通过调控RNA的表达来影响疾病的发生和发展,而不是在外显子中改变蛋白而影响表型。
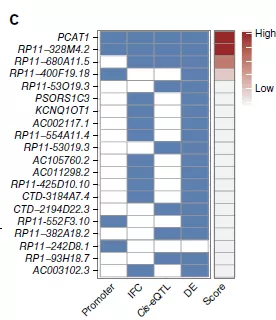
3. 使用TCGA数据库的SNP和表达数据,作者对target SNP做了cis-eQTL分析;用intercellular analysis (IFC),探索包含有risk SNP的enhancer和promoter的潜在相互作用;之后在结合差异基因的分析结果,作者对所包含的lncRNA进行打分,一共找到了45个PC的可能致病的lncRNA,其中PCAT1最为显著。作者之后的研究就都围绕着这个lncRNA进行的。
4. 作者首先通过实验证明了PCAT1和PC的关系,PC患者中这个lncRNA表达升高,减少PCAT1的表达,抑制了肿瘤的生长。和通常的lncRNA文章不同的是,作者没有继续关注这个lncRNA的功能了,而是把目光转向了SNP对这个lncRNA的调节上,大家可以学习一下。
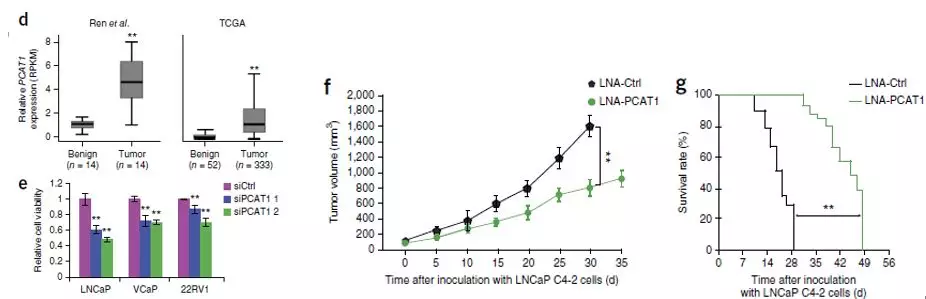
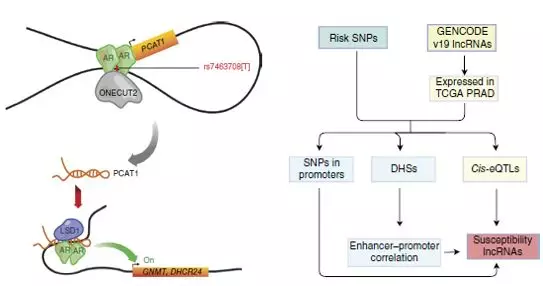
5. 之前的IFC分析表明,PCAT1的promoter区域和一个包含rs7463708的DHS区域存在潜在的相互作用,具有很强的关联,那么可能这个DHS所在区域就是PCAT1的enhancer。根据CHIP-Seq数据,这个区域可结合AR、FOXA1等转录因子。
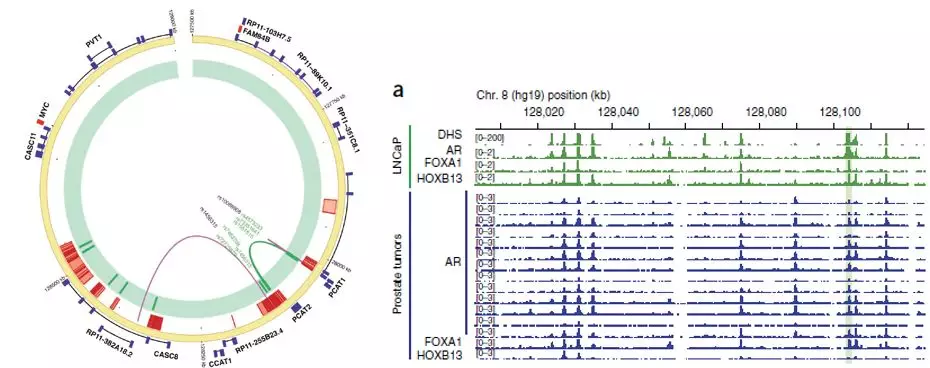
为了验证这个分析结果,作者就做了3C实验。确实发现了这两个区域的相互作用的信号,并且这个信号在前列腺癌细胞系的信号远远高于其他细胞系。更加有趣的是,也是作者太lucky,荧光报告实验中,携带risk allele T的rs7463078的enhancer功能强大,而non-risk allele G的enhancer和control相比并无区别。
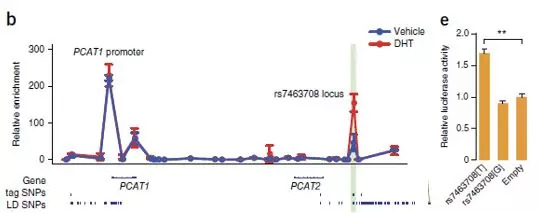
6. 对这一目标区域检查发现,在这个区域可以结合ONECUT2这个转录因子,并且实验证明这个转录因子更加青睐结合rs7463078的T allele的序列。在二氢睾酮(DHT)处理的细胞系中,AR和ONECUT2在这个区域的结合显著升高,当把ONECUT2敲出后,AR在这个区域的结合也显著降低。在之后的免疫共沉淀实验的结果也验证了他们之间的互作关系。PCAT1的表达在激素处理后也会增加,而knock down了ONECUT2,PCAT1的表达就下降了。
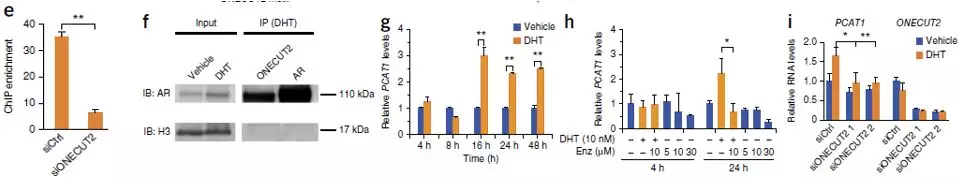
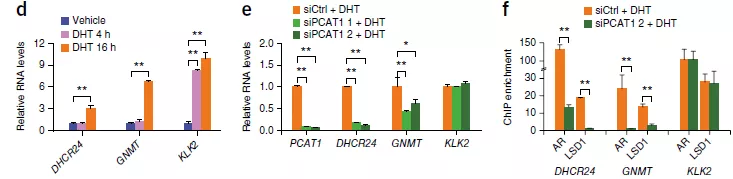
7. 对于上游机制的分析已经基本清楚,作者对于PCAT1的下游机制进行了探索。使用RNA Immunoprecipitation (RIP)和multidimensional protein identification technology (MudPIT)实验技术,作者发现PCAT1和AR以及LSD1会形成一个复合体,调节下游基因。这个复合体会特异性结合在GNMT、DHCR24的TSS上,调控下游基因表达。
总 结
这篇文章小编强烈推荐,不仅是因为作者巧妙的借助于公共数据库的挖掘,让读者学习到如何更加好的利用公共数据做自己的科研;更加是因为作者利用自己丰富的知识,拓宽了我们对于lncRNA的研究方法,做更多的尝试。