欢迎关注”生信修炼手册”!
GWAS ATLAS数据库收录了来自4756个人类不同表型的GWAS结果,并进行了不同表型间的遗传相关性分析,对应的文献发表在nature genetics上,链接如下
https://www.nature.com/articles/s41588-019-0481-0
数据库的网址如下
https://atlas.ctglab.nl/
该数据库不仅仅是一个gwas结果存储的数据库,对于收录的原始gwas结果,进行了深入挖掘,提供了risk loci, LDSC, MAGMA基因集关联分析,多种表型间的遗传相关性分析等结果。
分成了以下3大模块
1. Browse GWAS
查看gwas分析结果,包含了以下几点内容
manhattan plot
lead snp and risk loci
gene-based association results
SNP heritability and genetic correlations
GWAS分析最经典的展示结果就是曼哈顿图和QQ图了,示意如下
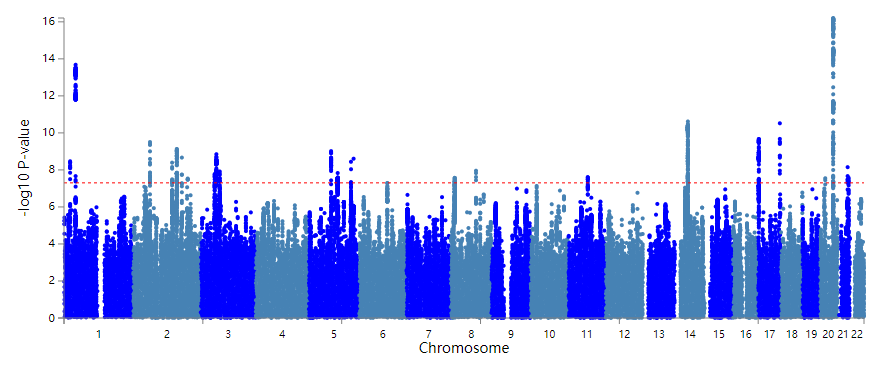
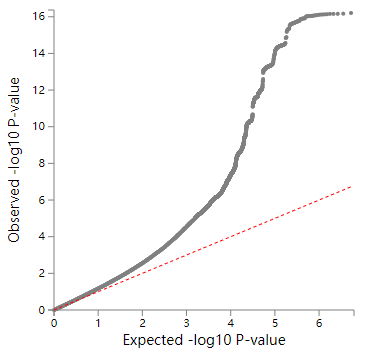
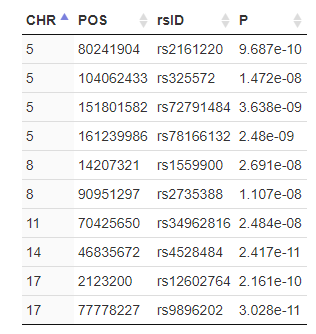
基于gwas结果,分析了lead snp和risk loci, 结果示意如下
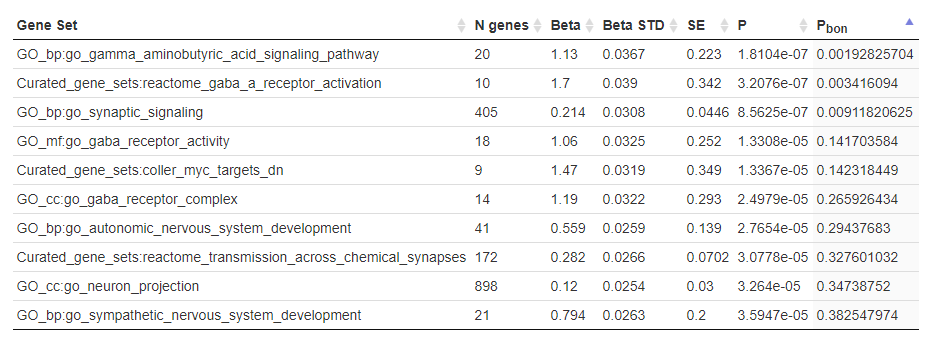
利用MAGMA软件进行了基因集的关联分析,结果示意如下
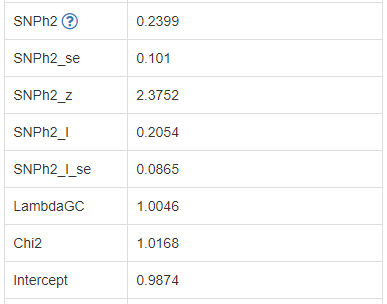
利用LDSC分析,评估了表型对应的SNP遗传力,结果示意如下
2. Multiple GWAS comparison
用于比较多个gwas分析结果,有两种展示形式,第一种是多个因子的相关性分析,示意如下
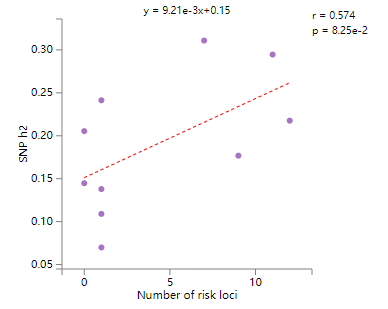
上图展示的是risk loci个数和snp遗传力之间的相关性,每个点表示一个表型的gwas结果。
第二种是热图,示意如下
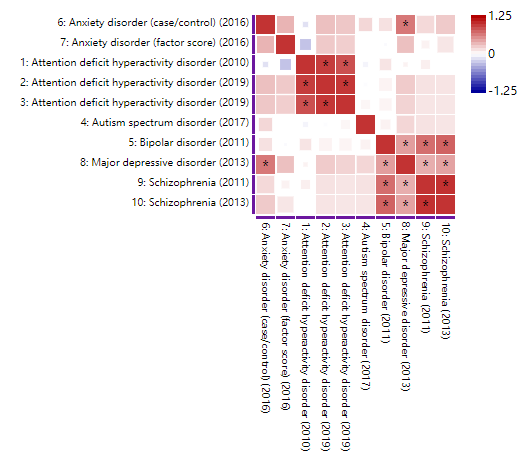
上图展示的是不同表型间遗传相似度的热图。
3. PheWAS
对于某个基因或者SNP位点,查看在不用表型中的关联分析结果,绘制如下所示的散点图
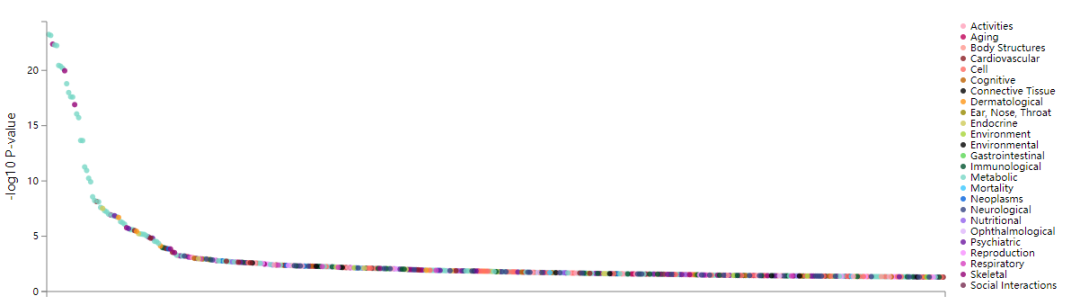
对应的表格数据如下
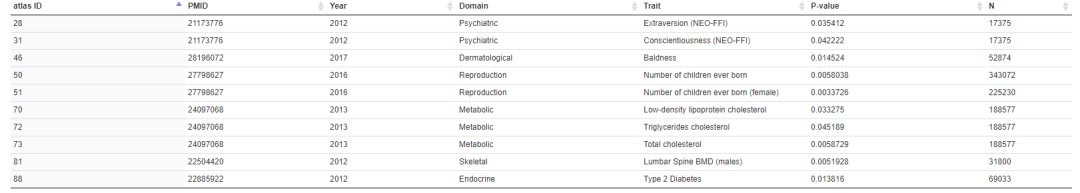
通过该数据库,不仅可以查看已有的gwas结果,更重要的是可以参考其后续挖掘的方法和思路。
·end·
—如果喜欢,快分享给你的朋友们吧—
往期精彩
3DIV:染色质空间互作数据库
扫描下方二维码,关注我们,解锁更多精彩内容!

生物信息入门
只差这一个
公众号
本文分享自微信公众号 - 生信修炼手册(shengxinxiulian)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
来源:oschina
链接:https://my.oschina.net/u/4580290/blog/4357099